[1036] RNA Métabolisme et pathogenèse bactérienne
→ Pseudomonas aeruginosa est une bactérie pathogène opportuniste qui se trouve dans de nombreuses niches écologiques et qui peut coloniser différents hôtes (mammifères et insectes). Naturellement très versatile, elle peut provoquer des infections aiguës et chroniques potentiellement mortelles chez l’homme, en particulier chez des personnes au système immunitaire fragilisé. La présence de P. aeruginosa dans les hôpitaux, où elle parvient à coloniser des dispositifs médicaux comme des respirateurs ou des cathéters, représente ainsi une sérieuse menace. En outre, sa capacité d’adaptation et sa résistance à un grand nombre d’antibiotiques rendent les infections à P. aeruginosa de plus en plus difficiles à traiter.
Dans notre groupe, nous étudions les mécanismes moléculaires qui coordonnent l’adaptation de P. aeruginosa à l’environnement et à l’hôte. Plus précisément, notre recherche se concentre sur le contrôle de l’expression des gènes effectué par des protéines et des complexes multiprotéiques liant l’ARN. Bien que ces complexes protéiques soient capables de réguler rapidement et de manière flexible l’expression des gènes, ils sont de manière surprenante peu étudiés chez les bactéries. Par leur liaison à des molécules d’ARN, ces régulateurs post-transcriptionnels affectent leur dégradation et/ou leur traduction en protéines. Notre laboratoire étudie ces régulateurs à travers deux approches :
A) Des études moléculaires et biochimiques qui identifient leur mécanisme d’action ;
B) Des études biologiques qui montrent leur importance pour la virulence et pathogenèse de la bactérie en utilisant la larve Galleria mellonella pour des études hôte-pathogène.
Nous espérons ainsi identifier des composants essentiels pour la pathogenèse de P. aeruginosa et de stimuler le développement de nouveaux médicaments qui les ciblent.
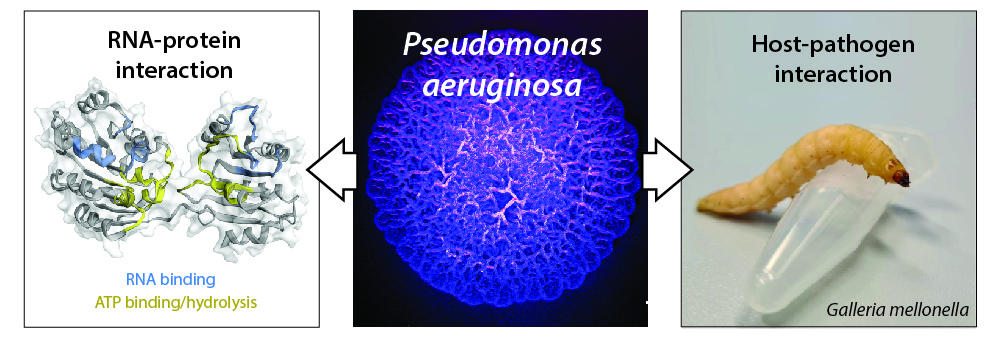
Figure légende : (panel au milieu) Surface d’une colonie biofilm de P. aeruginosa (stéréomicroscope, coloration artificielle) ; (panel gauche) model de la structure de l’ARN hélicase RhlE2 et les régions protéiques impliquées dans la liaison à l’ARN et dans la liaison/hydrolyse de l’ATP (Hausmann et al. 2021) ; (panel droite) photo d’une larve de Galleria mellonella, organisme respectent les principes 3R pour des études hôte-pathogène.