Physique et biologie explorent ensemble les mécanismes du vivant
Des scientifiques de l’UNIGE créent un modèle expliquant comment les protéines se répartissent de façon inégale dans les cellules. Un processus essentiel pour le développement des êtres vivants.
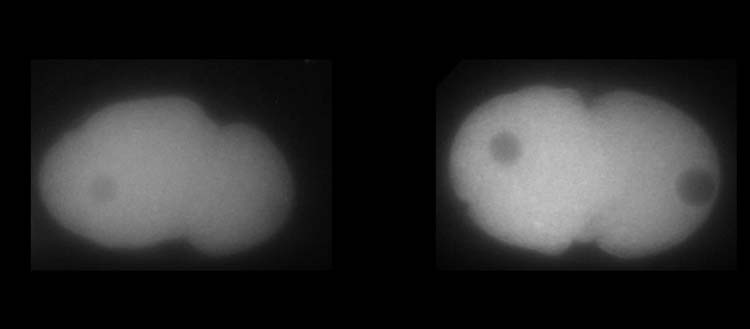
A gauche, la protéine MEX-5. A droite, la protéine PLK-1 dans des embryons de C. elegans. La couleur blanche montre la présence plus ou moins grande des protéines.
© UNIGE – Laboratoire Monica Gotta
Chacune de nos cellules contient environ 40 millions de protéines qui accomplissent ensemble toutes les tâches nécessaires à sa survie. Pour y parvenir, les différentes protéines doivent être concentrées en quantités spécifiques, au bon endroit au bon moment. Le processus qui permet le déroulement sans accrocs d’une distribution si précise se déroule à des résolutions spatiales si infimes que les outils standard de biologie cellulaire sont souvent incapables de le détecter. Pour comprendre comment fonctionne ce mécanisme, des scientifiques de l’Université de Genève (UNIGE) ont développé une approche novatrice qui combine des expériences de génétique et de biologie cellulaire à une modélisation faisant appel à la physique. Grâce à des algorithmes spécifiques, ils/elles ont simulé en 3D et sous forme dynamique la formation de gradients de protéines et ont pu expliquer ces phénomènes complexes. De plus, leur modèle peut être adapté à d’autres systèmes biologiques pour étudier la dynamique des protéines. Ces résultats sont à découvrir dans les Proceedings of the National Academy of Sciences.
Comme une goutte d’encre dans un verre d’eau, les protéines peuvent se diffuser et se répartir uniformément dans la cellule. Cependant, pour pouvoir accomplir certaines tâches, elles doivent former des gradients. «Les gradients résultent de la répartition inégale des protéines dans des zones spécifiques de la cellule; ils sont au cœur de nombreuses fonctions à l’échelle de la cellule et de l’organisme», explique Monica Gotta, professeure au Département de physiologie cellulaire et métabolisme et au Centre de recherche translationnelle en onco-hématologie (CRTOH) de la Faculté de médecine de l’UNIGE, qui a dirigé ces travaux. «Par exemple, les gradients de protéines sont importants pour la différenciation cellulaire, le processus par lequel les différents types de cellules qui constituent un organisme complexe peuvent émerger d’une unique cellule, l’œuf fécondé.»
Une utilisation du hasard
La protéine PLK-1, un régulateur clé de la division cellulaire, est plus concentrée sur le côté antérieur de l’embryon. Mais comment ce mécanisme peut-il se mettre en place, et quelles seraient les conséquences si le moindre détail venait à se dérégler? Les outils habituels de la biologie n’étant pas suffisants pour répondre à cette question, Monica Gotta a fait appel à une physicienne, Sofia Barbieri, chercheuse postdoctorale au Département de physiologie cellulaire et métabolisme de la Faculté de médecine de l’UNIGE. «En compilant tout ce qui était connu de ce processus biologique avec de nouvelles hypothèses, j’ai développé un modèle statistique de la formation des gradients de protéines basé sur les mathématiques probabilistes», explique Sofia Barbieri. «J’ai eu recours à des algorithmes de calcul spécifiques, appelés simulations de Monte-Carlo, du nom de la ville célèbre pour ses casinos.» Ces algorithmes sont utilisés pour modéliser des phénomènes d’un niveau de complexité élevé, comme la finance, la bourse ou la physique des particules.
L’équipe a ainsi pu simuler les gradients de protéines, non seulement en 3D, mais aussi dans le temps. Un tel modèle nécessitait cependant plusieurs allers-retours entre l’optimisation des paramètres et la comparaison avec les données biologiques: sa première version incorporait tous les éléments physiques et biologiques connus, puis des paramètres spécifiques ont été introduits pour tester plusieurs hypothèses concernant les variables inconnues. L’équipe a simulé ainsi les scénarios – possibles d’un point de vue physique et biologique – qui reproduisaient la dynamique des protéines permettant l’apparition du gradient. Ces scénarios étaient ensuite testés dans la réalité au travers d’expériences in vivo utilisant des embryons d’un petit ver, le nématode C. elegans (vidéo).
Des interactions protéiques complexes en jeu
Grâce à une constante interaction entre modélisation et biologie cellulaire, les scientifiques de l’UNIGE ont pu déterminer comment le gradient de PLK-1 s’établit et se maintient dans le temps. En effet, PLK-1 doit se lier et se délier activement de MEX-5, une autre protéine essentielle au développement des embryons de C. elegans, pour contrecarrer sa tendance naturelle à se diffuser de manière homogène dans la cellule. MEX-5 a en effet la capacité de modifier sa diffusion en fonction de sa position dans la cellule et d’interagir avec d’autres protéines, ce qui lui permet d’augmenter la quantité de PLK-1 aux endroits nécessaires. «Mais de manière assez surprenante, MEX-5 n’est pas si efficace dans sa tâche, car une grande quantité de PLK-1 n’est pas liée à MEX-5!» souligne Sofia Barbieri.
Cette étude fournit un modèle quantitatif unique pour comprendre les interactions dynamiques entre protéines et peut être adaptée à d’autres cellules ou protéines dont les mécanismes complexes ne peuvent être testés avec des expériences traditionnelles de biologie cellulaire. «Nos travaux démontrent que les collaborations interdisciplinaires sont de plus en plus importantes pour faire avancer la recherche», conclut Monica Gotta.
7 mars 2022